ارتباط بین سلامت واحد و میکروبیومهای انسان، حیوان و محیط زیست
ترجمه: دکتر سید شهرام میرزمانی
معاون بهداشت اداره بهداشت درمان درمان نیروی دریایی
DVM-MPH- Ph.D Candidate in Microbiology
Islamic Azad University, Science and Research Branch
ss.mirzamani@gmail.com
خلاصه
مفهوم سلامت واحد (One Health) بر روابط اکولوژیکی بین سلامت انسان، حیوان و محیط زیست تأکید می کند. بسیاری از مقالات سلامت واحد تا به امروز انتقال عوامل بیماری زا از حیوانات (برای مثال، زئونوزهای نوپدید) و محیط زیست را به انسان بررسی کرده اند.
توسعه سریع اخیر فناوری با توالی یابی DNA با توان عملیاتی بالا، این دیدگاه را گسترش داده و مطالعه کل جوامع میکروبی را در بر گرفته است.
استفاده از رویکرد سلامت واحد در میکروبیوم امکان در نظر گرفتن انتقال میکروب های بیماری زا و غیر بیماری زا را بین انسان، حیوان و محیط فراهم می کند.
در این مقاله مطالعات تحقیقاتی اخیر درباره چنین انتقالی، روش های مولکولی و آماری مورد استفاده و پیامدهای چنین روابط میکروبیومی بر سلامت انسان را بررسی می کند.
این مقاله مروری شواهدی را شناسایی کرده است که میکروبیوم محیط زیست و همچنین میکروبیوم حیوانات در تماس نزدیک باهم می توانند بر میکروبیوم انسان و پیامدهای سلامت انسان تأثیر بگذارند. چنین انتقال میکروبیوم می تواند در خانه و همچنین محیط کار اتفاق افتد.
شهرنشینی محیط های زیستی را ساخته است که منجر به تغییراتی در میکروبیوم محیط زیست شده است که می تواند عاملی در سلامت انسان باشد. در حالی که میکروبیوم انسان تحت تأثیر مواجهه محیط زیست قرار می گیرد، می تواند پاسخ به عوامل محیطی را نیز از طریق تأثیر بر عملکرد متابولیکی و ایمنی تنظیم نماید.
درک بهتر این تعاملات میکروبیوم بین انسان، حیوان و محیط زیست مشترک به توسعه مداوم رویکردهای مدل سازی آماری و اکولوژیکی بهبود یافته نیاز دارد. چنین درک پیشرفته ای می تواند منجر به مداخلات نوآورانه برای جلوگیری و مدیریت وضعیت ها یا حالات متنوع سلامتی و بیماری انسان شود.
مقدمه
میکروبیوم - از پاتوژن های منفرد تا جوامع میکروبی در حالی که میکروبیولوژی بالینی به طور سنتی بر نقش پاتوژن های فردی در بیماری انسان تمرکز دارد، دستیابی به موفقیت در تعیین توالی DNA با توان بالا (high throughput DNA sequencing) اکنون امکان مطالعه کل جوامع میکروبی را فراهم آورده است.
جوامع متنوع باکتری ها، آرکی ها و یوکاریوت های میکروبی که میکروبیوم انسان را تشکیل می دهند شامل ارگانیسم های غیر بیماری زای است که می تواند از طریق مکانیسم هایی مانند متابولیسم مواد مغذی و دارویی (1 ، 2)، سنتز ویتامین های ضروری (3)، دفاع در برابر عوامل بیماری زا (4)، فرآیند ثانویه اسیدهای صفراوی میزبان (5)، تنظیم سیستم ایمنی (6 ، 7)، مقاومت و حساسیت به عفونت (8) و حتی اصلاح رفتار (9) بر سلامتی انسان و هموستاز تأثیر بگذارد.
میکروبیوم ها در میان طیف سلامت واحد
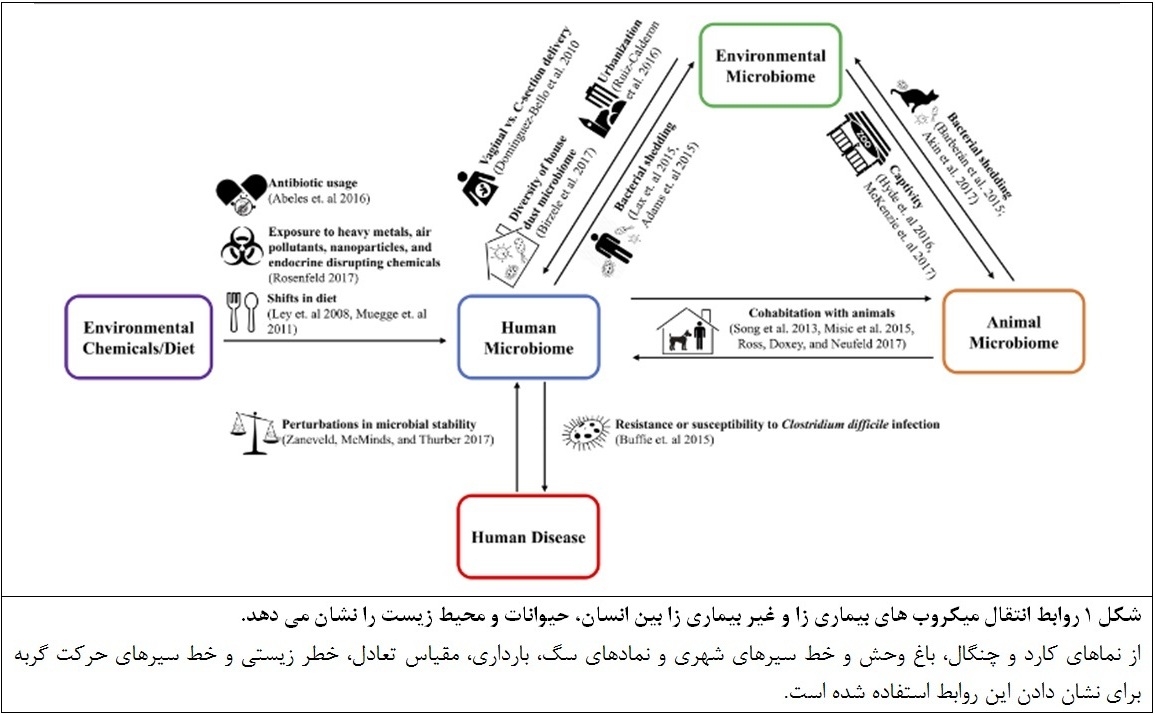
مفهوم سلامت واحد بر روابط اکولوژیکی بین سلامت انسان، حیوان و محیط زیست تأکید می کند (10). استفاده از رویکرد سلامت واحد در میکروبیوم مستلزم بررسی انتقال میکروب های بیماری زا و غیر بیماری زا بین انسان، حیوانات و محیط زیست است (11-13). این روابط انتقال در شکل 1 نشان داده شده است.
درک پیامدهای روابط میکروبیوم بین محیط و سلامت انسان و حیوانات ساکن در آن، پتانسیل رویکردهای نوآورانه و جامعی را در تشخیص، درمان و مداخله باز می کند (14).
گونه های مختلف حیوانات میکروبیوم های منحصر به فردی را در خود جای داده اند که در مقایسه با میکروبیوم انسانی اغلب دارای پیچیدگی برابر یا بیشتر هستند.
همانند انسان ها، میکروبیوم حیوانات بر سلامت دام، حیوانات خانگی، ناقلین بیماری و گونه های بنیادی (گونه هایی که در ساختن یک جامعه نقش پررنگی دارند) که اکوسیستم ها را حفظ می کنند، تأثیر می گذارد. برای مثال، میکروبیوم خوک ها میزان بروز بیماری های تنفسی آنها را نشان می دهد (15)، در حالی که میکروبیوم مرجان های سازنده صخره های مرجانی نقشی اساسی در پاسخ اکوسیستم های صخره به صید بیش از حد آنها، آلودگی مواد مغذی و گرم شدن کره زمین دارند (16).
به همین ترتیب، محیط های زیست میکروبیوم مشخصی دارند و این ممکن است بر سلامت انسان تأثیر بگذارد. برای مثال، موازات شهرنشینی آلرژی، آسم و سایر بیماری های مزمن در انسان افزایش یافته است (17) که احتمالاً مربوط به کاهش مواجهه با میکروارگانیسم های متنوع است (7).
درک ما از میکروبیوم های محیط زیست از طریق نمونه گیری در متروها (18–21)، آمبولانس (22)، سرویس بهداشتی (23)، کلاس های دانشگاه و ساختمان های اداری، و همچنین زیست بوم ها و زیستگاه های سراسر جهان (24) افزایش یافته است.
درک تأثیر سلامت انسان در مواجهه با میکروب ها از حیوانات یا محیط زیست، مستلزم تلفیق چارچوب ارزیابی مواجهه با مدل های پیش بینی کننده اکولوژی میکروبی و پیامدهای سلامتی مرتبط با آن است.
شواهد موجود نشان می دهد که مونتاژ میکروبیوم انسان و سایر حیوانات توسط چندین فرآیندی از جمله مواجهه با میکروب ها (برای مثال، از والدین، افراد دیگر، حیوانات و محیط زیست)؛ پایش و پالایش مواجهه میکروبی بر اساس تعامل صفات میکروبی و میزبان (برای مثال، متابولیسم و ایمنی بدن)؛ و پیامد میان کنش های رقابتی یا همکاری بین میکروب ها و فاژها در محیط زیست میزبان تحت تأثیر قرار می گیرد.
در این مطالعه مروری کوتاه، محققان درباره نکات کلیدی رابطه متقابل این فرآیندها بحث می کنند، و بر دانش فعلی درباره چگونگی انتقال میکروبی بین انسان، حیوانات و محیط زیست بر سلامتی انسان تأکید می نمایند. سپس آنها چالش های اصلی روش شناختی و راه حل های بالقوه برای درک پیش بینی کننده این فرایندها را ترسیم می نمایند.
جنبه های کلیدی رابطه بین سلامت واحد و میکروبیوم
آثار میکروبیوم محیط زیست بر میکروبیوم انسانی
میکروبیوم انسان می تواند در اثر تماس با سطوح، حیوانات (25 ، 26) و سایر افراد از طریق بلع غذا و آب؛ یا استنشاق (27 ، 28) ایجاد شود. برعکس، میکروبیوتای دستگاه گوارش همچنین ممکن است با تغییر در ایمنی ذاتی و اکتسابی و از طریق اثرات بر روی مخاط دستگاه گوارش، در برابر مواجهه محیطی با انگل ها و سایر عوامل بیماری زا مقاومت کند. (29)
تصور می شود که آثار مواجهه میکروبی در اوایل زندگی آغاز می شود. نشان داده شده که زایمان واژینال در مقابل سزارین بر بذرپاشی و جوانه زایی میکروبی دستگاه گوارش نوزادان تأثیر می گذارد (30). این تغییر در ترکیب میکروبی در اوایل زندگی، پیامدهای بالقوه سلامتی را در توسعه سیستم ایمنی بدن با تأثیرگذاری بر سلامت از جمله رینیت آلرژیک، آسم، بیماری سلیاک، دیابت و گاستروانتریت به همراه دارد (31).
محیط زیست ساخته شده می تواند منبعی برای انتقال میکروبی به ساکنان آن باشد و هرچه محیط زیست بسته تر باشد، پتانسیل بیشتری برای این انتقال وجود دارد (12 ، 32).با مطالعه اژدهای کومودو اسیر میکروبیوم بین محیط زیست محصور و میکروبیوم های مدفوعی، بزاقی و پوستی اژدهای کومودو (Komodo dragons) این را شناسایی کرده است (12). همان مطالعه نشان داد که بیشترین تنوع بتا (تفاوت در ترکیب گونه ها) بین میکروب های انسان و خانه های آنها از اژدهای کومودو و محیط محصور آنها بیشتر است، و این نشان می دهد که زندگی با اطمینان و ایمنی بیشتر می تواند با تأثیر بیشتر محیط زیست ساخته شده بر میکروبیوم میزبان همراه باشد (12) .
پرورش در اسارت در مقابل پرورش وحشی با کاهش تنوع بتا در جامعه باکتریایی در طیف وسیعی از گونه های جانوری همراه بوده است (33). برای مثال، قرار دادن دوزیستان وحشی (34 ، 35) در محیط های محصور، از تنوع باکتریایی بر روی پوست آنها می کاهد، مگر اینکه آنها با یک بستر خاک از زیستگاه های طبیعی خود نگهداری شوند.
شبیه به اسارت در حیوانات، شهرنشینی جمعیت های انسانی منجر به ایجاد محیط های بسته تر و رژیم های غذایی با تنوع کمتر می شود که می تواند دارای اثرات میکروبیوم باشد (36). میکروبیوم خانگی در دهکده های روستایی با محیط شهری بسیار متفاوت است (36). به همین ترتیب، میکروبیوم خانه های پرورش دهندگان خوک از تنوع و فراوانی میکروبی بیشتری در مقایسه با خانه های حومه های شهر برخوردار است (37).
میکروبیوم محیط کار همچنین می تواند میکروبیوم انسانی را تحت تأثیر قرار دهد. در تحقیقی که بر روی کارگران در آزمایشگاه های تحقیقات حیوانات انجام شد، تجزیه و تحلیل ردیابی منبع (Source Tracker analysis) (روشی برای استنباط منابع میکروبی) نشان داد که میکروبیوم محیط کار به میکروبیوم دهان، بینی و پوست کارگران کمک می کند (38).
به اشتراک گذاشتن میکروبیوم بین حیوانات و انسان ها
عناصر جوامع میکروبی می توانند از طریق تماس نزدیک بین انسان و حیوان منتقل شوند.
مطالعه ای در مورد میکروبیوتای پوستی زوج های مشترک نشان داد که علاوه بر اشتراک میکروبی فرد به فرد، صاحب حیوانات خانگی با تنوع بیشتر میکروبیوم پوست ارتباط دارد (39).
مطالعات دیگر نشان داده است که میکروبیوم بین انسان و سگ ها در یک خانه، به ویژه میکروبیوتای پوستی به اشتراک گذاشته می شود (11).
علاوه بر این، زوج های مشترک که صاحب سگ بودند، بیشتر از سایر زوج های فاقد سگ باکتری های پوستی مشترک داشتند که این احتمالاً به دلیل انتقال اضافی از طریق وسایل یک حیوان خانگی است (11).
مطالعه دیگری نشان داد که افرادی که در یک خانه با حیوانات خانگی زندگی می کنند در مقایسه با افرادی که حیوانات خانگی ندارند، شباهت بیشتری میکروبیوم بینی و پوست آنها دارند، که تأثیر حیوانات خانگی بر تقویت تبادل میکروبی را نشان می دهد (40).
الگوهای مشابهی در به اشتراک گذاشتن میکروب ها بین انسان و دام های همجوار آنها گزارش شده است. مشخص شده که پرورش خوک تأثیر مهمی بر میکروبیوم بینی پرورش دهندگان خوک دارد (41).
یک مطالعه در مورد کودکان روستاهای کنیا که در تماس نزدیک با دام ارتباط داشتند، نشان داد که در حالی که بیشترین میزان تشابه میکروبیوم روده بین خواهر و برادر در یک خانوار وجود دارد، در برخی از خانه ها شواهدی مبنی بر به اشتراک گذاشتن اجزای میکروبیوم بین کودکان و گاوهای همجوار آنها وجود داشت (42).
اثرات انسانی و حیوانی بر میکروبیوم محیط زیست
منبع-مخزن مدل های اکولوژیکی (Source-sink ecological models ) بر اساس این مفهوم است که زیستگاه های با کیفیت بالا (منابع) وجود دارد که موجودات در آن رشد می کنند و زیستگاه های با کیفیت پایین تر (مخازن) که موجودات اضافی از این منبع (source) خارج می شوند (13).
شواهدی وجود دارد که انسان و حیوانات می توانند منبعی از میکروب ها باشند که به سمت مخزن (sink) محیط زیست خود حرکت می کنند.
در محیط های شهری و چهار دیواری ساخته شده، در مقایسه با سکونتگاه های روستایی و باز، محتوای بیشتری از میکروب های مرتبط با انسان وجود دارد (36).
ورود حیوانات خانگی به یک خانوار می تواند منجر به تغییرات قابل توجهی در میکروبیوم گرد و غبار خانه شود (43 ، 44).
اغلب این کلنی زایی مخازن محیطی توسط انسان و حیوان سریع است و امضای میکروبی محیطی گذرا است مگر اینکه ریزش و حضور مداوم منبع وجود داشته باشد (13 ، 45)
میان کنش های مواد شیمیایی و پاتوژن محیطی با میکروبیوم انسانی
مواجهه با مواد شیمیایی در محیط می تواند تغییرات دیسبیوزی را در ترکیب میکروبیوم روده ایجاد کند یا فعالیت متابولیکی میکروبیوتای روده را تغییر دهد (46). برعکس، تفاوت در ترکیب میکروبیوتای روده می تواند نحوه متابولیسم سموم محیطی را تغییر دهد (46).
ارتباط سلامت و سازوکارهای این میان کنش ها، زمینه های فعال برای تحقیق هستند (47 ، 48).
مصرف آنتی بیوتیک به طور قابل توجهی تنوع و فراوانی میکروبیوم روده انسان را کاهش می دهد (49). از طریق خانواده های آنزیمی مانند آزورداکتازها، نیتروردوکتازها، β -گلوکورونیدازها، سولفاتازها و β -لیازها (46)، میکروارگانیسم های روده می توانند متابولیسم زنوبیوتیک (xenobiotic metabolism) را تحت تأثیر قرار دهند.
باکتری های دستگاه گوارش همچنین می توانند انواع مواد شیمیایی محیطی مانند آفت کش های هیدروکربن های معطر چند حلقه ای (PAH) ، پلی کلروبی فنیل ها، مشتقات بنزن، ملامین، شیرین کننده های صنعتی و فلزات را متابولیزه کنند (46)، سمیت آنها را تغییر می دهند (50-52).
میکروبیوم های دیگر در کنار دستگاه گوارش ممکن است پاسخ به مواد مسموم کننده محیطی را تنظیم کنند.
مطالعات انجام شده بر روی جوندگان نشان می دهد که میکروبیوم تنفسی، با تولید اسیدهای چرب با زنجیره کوتاه (SCFA)، می تواند پاسخ مجاری تنفسی را به مواجهه با ازن تنظیم نماید (53)
پیامدهای انتقال میکروبی غیر بیماری زا برای سلامت انسان
در حالی که پیامدهای انتقال میکروب های بیماری زا آشکار است، در مقالات رو به رشد نیز پیامدهای سلامتی انتقال میکروب های غیر بیماری زا بین انسان، حیوانات و محیط وجود دارد.
در نوزادانی که مواجهه میکروبی کمتری داشته اند، مانند افرادی که تولد سزارین را تجربه کرده اند و در محیط زیست های شهری زندگی می کنند، در مقایسه با کودکانی که در مزارع رشد می کنند، خطر آسم و آلرژی بالاتری دارند و که این یافته ها از "فرضیه بهداشت" حمایت می کنند (54).
مشخص شده كه ميكروبيوم نمونه هاي گرد و غبار بين خانه هاي كودكان مبتلا به آسم و بدون آسم تفاوت دارد (27)، و تنوع جامعه ميكروب هاي گرد و غبار در خانه مي تواند پيش بيني كننده وضعيت آسم در كودكان ساكن در آن خانه باشد (28).
مدل های موش گنوتوبیوتیک (Gnotobiotic mouse models) از تأثیر عللی انتقال میکروب های غیر بیماری زا بر سلامت میزبان پشتیبانی می کنند. برای مثال، انتقال مدفوع انسان های چاق به موش های گنوتوبیوتیک باعث افزایش وزن نسبت به موش های گنوتوبیوتیک پیوند داده شده با مدفوع انسان لاغر می شود (55).
در ترکیب با اثرات ترکیب میکروبیوم بر متابولیسم داروها و سموم محیطی، این مشاهدات نشان می دهد که انتقال میکروب های غیر بیماری زا می تواند پیامدهای قابل توجهی برای سلامتی داشته باشد.
ابزارهای آماری تجزیه و تحلیل روابط میکروبیومی سلامت واحد
کشف میان کنش های بین میکروبیوم های انسانی، حیوانی و محیطی
رویکردهای سلامت واحد هدف درک تأثیرات متقابل میکروبیوم های انسانی، حیوانی و محیط زیست بر روی یکدیگر به منظور طراحی نهایی مداخلات برای بهبود سلامت انسان است.
اولین قدم برای انجام این کار، توصیف نحوه تغییر فاکتور مورد علاقه در جوامع میکروبی و سلامت انسان است.
مطالعات پیگیری که مستقیماً میکروبیوم را دستکاری می کنند (برای مثال، در موش های گنوتوبیوتیک) می توانند کمک کنند تا بررسی کنیم آیا تغییرات میکروبیوم مشاهده شده نقش علیتی دارند یا پیامدهای ثانویه تغییر وضعیت سلامتی را نشان می دهند.
پس از جمع آوری نمونه و استخراج DNA میکروبی، توالی یابی ژن نشانگر ، ژن مورد نظر را تکثیر می كند و سپس آن را توالی یابی می کنند تا ترکیب فیلوژنی و طبقه بندی میکروبیوم را استنباط کند.
ژن هایی که معمولاً مورد هدف قرار می گیرند شامل ژن RS ریبوزومی ( 16S rRNA ) برای مطالعات باکتری ها و آرکی ها، نواحی ITS2 برای بررسی قارچ ها یا ژن 18S rRNA برای یوکاریوت های میکروبی هستند.
در مقابل، قطعات و توالی متاژنومیکس تفنگی (shotgun metagenomics ) کل DNA میکروبی موجود را مورد بررسی قرار می دهد، و سپس مجموعه توالی ها را تجزیه و تحلیل می کند تا نه تنها گونه های باکتریایی، بلکه وجود یا عدم وجود و انواع ژن های خاص را نیز شناسایی کند (56 ، 57).
هنگامی که بطور عمیق توالی یابی با توانایی بسیار بالا انجام می شود، متاژنومیکس تفنگی همچنین می تواند ژنوم های میکروبی جزئی را با استفاده از ترکیب بندی ترکیبی یا همبستگی در ژن های متعلق به همان ارگانیسم در بین نمونه ها بازیابی کند (58).
از هر فناوری که استفاده شود، طراحی دقیق نمونه گیری است که فرضیه های خاص مورد آزمایش را برطرف می کند و جمع آوری ابرداده ها کافی (برای مثال در مورد متغیرهای مخدوش کننده) برای تفسیرپذیری نتایج ضروری است (59).
سئوالات متداولی كه مي توان از طريق رویکردهای توالي ميكروبيوم، همراه با آزمايشات هدفمند، برطرف كرد، شامل موارد زير است: چگونه هر یک از این عوامل غنا (richness ) و یکنواختی (evenness) (60-62)، ترکیب (63-66)، پایداری و عملکرد میکروبیوم را تغییر می دهد؛ منبع (67)، جهت و میزان انتقال میکروبی؛ و کدام گونه های میکروبی (یا کنسرسیوم های گونه ای) میانجی پیامدهای کلیدی سلامت هستند (جعبه 1).
به سمت مدل های پیش بینی کننده انتقال میکروبی
پیش بینی و دستکاری بالقوه انتقال میکروب ها بین انسان، حیوان و محیط زیست بستگی به درک عواملی دارد که باعث انتقال یا جلوگیری از انتقال می شود. این هدف به موازات نقش مدل های پیش بینی کننده - مانند مدل مواجهه انسانی - در علم مواجهه (exposure science)، با این عوارض اضافی که مواجهه مورد بحث موجودات بیولوژیکی با پویایی های پیچیده اکولوژیکی و تکاملی آنها قرار دارد.
تجزیه و تحلیل گونه های نشانگر یک روش کلیدی برای تشخیص منبع آلودگی میکروبی سایت های موجود در محیط زیست بوده است (87). با این حال، روش های گونه های نشانگر به طور معمول فرض می کنند که گونه هایی منحصر به فردی برای نمونه های منبع در مقابل مخزن وجود دارد.
مدل های چند جمله ای دیریکله (Dirichlet-Multinomial Model ) یک رویکرد جایگزین است که سعی دارد جوامع مشاهده شده میکروبی را به عنوان مخلوطی از پروفایل های میکروبی از جوامع منبع مختلف توضیح دهد، همانطور که به هنگام انتقال میکروب ها اتفاق می افتد. برای مثال، نرم افزار ردیابی منبع (SourceTracker software) برای تخمین نسبت میکروبیوم خاص متشکل از میکروب های ناشی از یک منبع مشخص (88) استفاده می شود.
با این حال، از آنجا که انتقال میکروبی می تواند در یک سیستم محصور در گردش باشد، مجموعه داده های طولی یا آزمایش های اضافی اغلب برای ایجاد جهت و پویایی انتقال میکروبی مورد نیاز است. لاکس و همکاران این مسئله را با استفاده از ردیابی منبع به صورت طولی برای بررسی انتقال میکروب ها بین هفت خانواده و خانه های آنها در طول دوره های اقامت مداوم در مقابل حرکت حل کرد (45). آنها علاوه بر تجزیه و تحلیل ردیابی منبع خود را با شبکه های پویای بیزی (dynamic Bayesian networks ) برای آزمایش جهت انتقال میکروبی و تأیید اینکه انسان ها بیشتر از سطوح فیزیکی منبع باکتریایی هستند، آزمایش کردند. یک محدودیت ردیابی منبع این است که تمام نمونه های منبع به طور جداگانه در نظر گرفته می شوند - وابستگی متقابل بین محیط های منبع مدل سازی نمی شود. یک راه حل که در حال حاضر توسط بسته هایی مانند BioMiCo در حال بررسی است، مدل سازی محیط های زیست منبع به صورت سلسله مراتبی است (89). همه این روش ها هنوز فرض می کنند که انتقال آنی بوده است. پیش بینی دقیق پیامدهای انتقال میکروبیوم در طول زمان، یا تشخیص تغییرات میکروبیوم به دلیل انتقال گذشته، احتمالاً به مدل های دقیق تر و محیطی بیشتری احتیاج دارد که دارای میان کنش های میکروب-میکروب و میکروب- فاژ هستند.

مفهوم اکولوژیکی گونههای کلیدی (keystone species) پیش بینی می کند که گونه های خاصی می توانند تأثیرات زیادی روی شبکه های متقابل میکروبی (67 ، 90 ، 91)، از جمله میکروبیوم روده (92 ، 93) داشته باشند. با این حال میان کنش های پیچیده ای در سیستم های اکولوژیکی شناسایی و اعتبار سنجی حضور و نقش این گونه های کلیدی را با مشکل مواجه می کند (93).
مدل های تعاملات شکارچی-طعمه لوتکا-ولترا (Lotka-Volterra models of predator-prey interactions) یکی از روش های استنباطی میان کنش های رقابتی و همکاری در جوامع میکروبی است (94). چارچوب لوتکا-ولترا تعمیم یافته پویایی تعداد دلخواه گونه ها را مدل سازی می کند و می تواند با استفاده از ترکیبی از یادگیری ماشینی و مدل سازی اکولوژیکی برای استنباط شبکه های میکروبی زمینه ای و ایجاد آشفتگی های وابسته به زمان گسترش یابد (94). رویکردهای داده محور مانند چارچوب لوتکا-ولترا تعمیم یافته از داده های سری زمانی به خوبی بهره نمی گیرند و این امر بر نیاز به مطالعات طولی تأکید می کند.
ایجاد علیت و نه فقط همبستگی همچنان یک چالش اصلی برای تحقیقات میکروبیوم مرتبط با سلامت واحد خواهد بود. برای مثال، میان کنش های میکروبی رقابتی یا مشارکتی که از طریق مدل سازی استنباط می شوند می توانند به صورت آزمایشی (در شرایط آزمایشگاهی یا موجودات زنده مدل) آزمایش شوند.
چندین پیشرفت اخیر از بینش های بوم شناختی برای پیش بینی پاسخ میکروبیوم ها استفاده کرده اند. برای مثال، درک نقش رقابت در جایگاه میکروبی به کرنی و همکاران اجازه داد تا با تطبیق سویه های شناخته شده توانایی متابولیکی در ساخت یک آشیان (niche) از طریق افزودن جلبک دریایی به رژیم غذایی، یک میکروب را در روده انسان مهندسی کنند (95)
پیش بینی رقابت یا همکاری میکروبی - بر اساس الگوهای مثبت یا منفی همزمانی - با موفقیت پیش بینی کرد که کدام میکروب ها باعث افزایش تنوع آلفا در نماتوستلا وکتنسیس (Nematostella vectensis) می شوند (96).
رویکرد مشابهی در پیش بینی عفونت های ویروسی قابل انتقال بین انسان و حیوان استفاده شده است. عوامل خطر برای نوپدیدی عفونت ویروسی قابل انتقال بین انسان و حیوان از میزبان های پستانداران شامل ارتباط فیلوژنتیکی میزبان با انسان، طبقه بندی حیوانات (یک نماینده برای صفات) و دامنه همپوشانی با جمعیت های انسانی (احتمالاً یک نماینده برای مواجهه) می شود (97).
چالش های روش شناختی خاص برای رویکردهای سلامت واحد
از آنجا که رویکردهای سلامت واحد اغلب تغییرات میکروبیوم های انسانی را با چندین کنشگرهای حیوانات یا محیط زیست مقایسه می کند، چندین چالش روش شناختی خاص بوجود می آید که ممکن است در مطالعات صرفاً بالینی اعمال نشود.
یکی از چالش های مطالعات میکروبیوم یکپارچه انسان، حیوانات و محیط زیست، از هم گسیختگی نقش ژنتیک میزبان در پویایی انتقال میکروبی و پیامدهای سلامتی آن خواهد بود.
در بین بیماران انسانی، تفاوت های ژنتیکی به عنوان عوامل مهمی که میکروبیوتای روده را شکل می دهند، شناخته شده اند (98). حساسیت به انتقال میکروبی ممکن است مربوط به عوامل ژنتیکی میزبان باشد که بر وجود یا عدم وجود سویه های میکروبی تأثیر می گذارد. ژنتیک میزبان همچنین ممکن است به طور بالقوه بر پیامدهای بالقوه سلامتی انتقال های میکروبی که اتفاق می افتد، تأثیر بگذارد. بدین ترتیب، مطالعات آینده که نقش ژنتیک میزبان را در تنظیم پویایی انتقال مورد بررسی قرار دهند، راه های امیدوارکننده برای تحقیق هستند.
با این وجود، نقش ژنتیک میزبان هنگام مقایسه نژادهای حیوانات اهلی یا بین گونه های جانوری، احتمالاً بسیار بیشتر است. تجزیه و تحلیل اخیر عفونت های ویروسی قابل انتقال بین انسان و حیوان نشان داد که خفاش ها یک ناقل مهم اختصاصی هستند (97). با وجود این که بین تقریبا 950 تا 1250 گونه خفاش در سراسر جهان وجود دارد، هرکدام تنوع غذایی، استراتژی تاریخچه حیات، درجه همپوشانی با سکونتگاه های انسانی و میزان تنوع ژنتیکی درون گونه ای خود را دارند.
بنابراین، رویکردهای سلامت واحد از مطالعات اکولوژیکی و تکاملی بهره مند می شوند که تنوع طبیعی تعامل میکروب میزبان را به تعداد قابل قبول تری از قوانین کلی تقسیم کنند. برای مثال، گروه های بسیاری از گروه های جانوری و گیاهی تشابهاتی بین میکروبیوم های گونه ای تا حد قابل توجهی در مسیر تکاملی یا فیلوسیمبیوز (phylosymbiosis) (99) از خود نشان می دهند، که در آن ترکیب کلی جوامع میکروبی بین گونه ای از ساختار درخت فیلوژنتیکی که میزبانان آنها را بهم مرتبط می سازد، پیروی می کنند.
گونه های جانوری با ساختار شبیه به درخت تکامل مرتبط هستند. در مقایسه میکروبیوم ها بین گونه های مختلف جانوری، اصلاح وابستگی فیلوژنتیکی با استفاده از روش های مقایسه ای فیلوژنتیکی ضروری است (100). مقایسه هر صفت، از جمله ترکیب میکروبیوم، در بین گونه های جانوری با استفاده از روش های آماری سنتی، ساده فیلوژنتیکی در بین گونه ها، تلویحاً فرض می کند که همه گونه ها به طور یکسان با هم مرتبط هستند ("فیلوژنی ستاره ای (star phylogeny)"). این می تواند باعث ایجاد میزان بسیار بالای مثبت کاذب شود (100).
اگر به طور اتفاقی دو صفت یکبار به طور تصادفی با هم پدیدار شوند، اما تبارهایی که در آن به وجود آمده اند به طور گسترده بیان کنند، به نظر می رسد که این صفات کاملا با هم ارتباط دارند، حتی اگر این دو صفت هیچ گونه ارتباط علیتی با یکدیگر نداشته باشند. این مسئله در اکولوژی و تکامل به خوبی درک شده است و می توان با استفاده از روش هایی مانند تضادهای مستقل فیلوژنتیکی و حداقل مربعات تعمیم یافته فیلوژنتیک (PGLS ) (phylogenetic generalized least squares ) این مشکل را برطرف کرد. تعدادی از بسته های R این روش ها را اجرا می کنند، از جمله ایپ (101)، فانگورن (102)، فیتول (103)، پیکانت (104)، کپر (105)، گایگر (106) و فیلوم (107). ملاحظات مشابهی هنگام مقایسه ویژگی ها – مانند دامنه ژن های زمینه میزبان - در پاتوژن ها یا همزیست های مختلف میکروبی وجود دارد (108).
مسیرها و چالش های آینده
در انسان، پیوندهای مدفوع نوید بالینی قابل توجهی برای درمان عفونت مکرر کلستریدیوم دیفیستیل می دهد (109).
درک بهتر پیامدهای سلامتی ترکیب بندی مختلف میکروبیوم ها در انسان و حیوانات می تواند به همین ترتیب فرصت هایی را برای مداخلات بالینی فراهم نماید که میکروبیوم را مستقیماً اصلاح کند و همچنین مداخلات سیاست های بهداشتی را بطور غیرمستقیم اصلاح نماید.
با در دسترس بودن روزافزون داده های اومیکس، ارزیابی خطرات بهداشتی و منافع ناشی از روابط خاص میکروبیوم به مدل های جدید پیش بینی کننده ای نیاز خواهد بود که برهمکنش های پیچیده میکروبیوم را در نظر بگیرید (110).
تلفیق مدل های زیست محیطی برای پیش بینی رفتار اکوسیستم در چنین ارزیابی خطری ممکن است قابلیت پیش بینی را افزایش دهد. این می تواند شامل ارزیابی و اصلاح میکروبیوم محیط ساخته شده برای افزایش سلامت انسان و حیوانات در خانه باشد.
امکان دیگر تنظیم میکروبیوم های حیوانات همراه (companion animals) از طریق رژیم غذایی یا پروبیوتیک ها است تا تأثیر آنها بر سلامتی انسان های زیستگاه مشترک تأثیر بگذارد.
چنین امکاناتی برای مداخله در این مرحله همچنان در حد حدس و گمان باقی می ماند، اما هدف از تحقیق میکروبیوم سلامت واحد باید تعریف همزیستی سالم و مداخلات آزمایشی برای بهینه سازی تبادل میکروبیوم بین انسان، حیوانات و محیط زیستی باشد که آنها با هم به اشتراک می گذارند.
منبع (اصلی):
Trinh P, Zaneveld JR, Safranek S and Rabinowitz PM ( 2018) One Health Relationships Between Human , Animal, and Environmental Microbiomes: A Mini-Review . Front. Public Health 6:235. doi: 10.3389/fpubh. 2018.00235
REFERENCES
1 . Wallace BD, Wang H, Lane KT, Scott JE, Orans J, Koo S, et al. Alleviating cancer drug toxicity by inhibiting a bacterial enzyme. Science (2010) 330:831 – 5 . doi: 10.1126/science.1191175
2 . de Clercq NC, Groen AK, Romijn JA, Nieuwdorp M. Gut microbiota in obesity and undernutrition. Adv Nutr An Int Rev J. (2016) 7:1080–9 . doi: 10.3945/an.116.012914
3 . Kau AL, Ahern PP, Griffin NW, Goodman AL, Jeffrey I. Human nutrition , the gut microbiome, and immune system: envisioning the future. Nature (2012) 474:327–36. doi: 10.1038/nature10213
4 . Abt MC, Pamer EG. Commensal bacteria mediated defenses against pathogens. Curr Opin Immunol. (2014) 29:16–22. doi: 10.1016/j.coi . 2014.03.003
5 . Wahlström A, Sayin SI, Marschall HU, Bäckhed F. Intestinal crosstalk between bile acids and microbiota and its impact on host metabolism. Cell Metab. (2016) 24:41–50. doi: 10.1016/j.cmet.2016.05.005
6 . Round JL, Mazmanian SK. The gut microbiome shapes intestinal immune responses during health and disease. Nat Rev Immunol. (2009) 9:313–23 . doi: 10.1038/nri2515
7 . Belkaid Y, Hand TW. Role of the microbiota in immunity and inflammation . Cell (2014) 157:121–41. doi: 10.1016/j.cell.2014.03.011
8 . Buffie CG, Bucci V, Stein RR, McKenney PT, Ling L, Gobourne A , et al. Precision microbiome reconstitution restores bile acid mediated resistance to Clostridium difficile. Nature (2015) 517:205–8. doi: 10.1038 / nature13828
9 . Dinan TG, Stilling RM, Stanton C, Cryan JF. Collective unconscious : how gut microbes shape human behavior. J Psychiatr Res. (2015) 63:1–9 . doi: 10.1016/j.jpsychires.2015.02.021
10 . Davis MF, Rankin SC, Schurer JM, Cole S, Conti L, Rabinowitz P, et al . Checklist for one health epidemiological reporting of evidence (COHERE) . One Health (2017) 4:14–21. doi: 10.1016/j.onehlt.2017.07.001
11 . Song SJ, Lauber C, Costello EK, Lozupone CA, Humphrey G, BergLyons D, et al. Cohabiting family members share microbiota with one another and with their dogs. Elife (2013) 2: e00458. doi: 10.7554 / eLife.00458
12 . Hyde ER, Navas-Molina JA, Song SJ, Kueneman JG, Ackermann G, Cardona C, et al. The oral and skin microbiomes of captive komodo dragons are significantly shared with their habitat. mSystems (2016) 1: e00046-16 . doi: 10.1128/mSystems.00046-16
13 . Adams RI, Bateman AC, Bik HM, Meadow JF. Microbiota of the indoor environment: a meta-analysis. Microbiome (2015) 3:49 . doi: 10.1186/s40168-015-0108-3
14 . Flandroy L, Poutahidis T, Berg G, Clarke G, Dao M-C, Decaestecker E, et al . The impact of human activities and lifestyles on the interlinked microbiota and health of humans and of ecosystems. Sci Total Environ. (2018) 627:1018 –
38 . doi: 10.1016/j.scitotenv.2018.01.288
15 . Niederwerder MC. Role of the microbiome in swine respiratory disease. Vet Microbiol. (2017) 209:97–106. doi: 10.1016/j.vetmic.2017.02.017
16 . Webster NS, Reusch TBH. Microbial contributions to the persistence of coral reefs. ISME J. (2017) 11:2167–74. doi: 10.1038/ismej.2017.66
17 . Asher MI. Urbanisation, asthma and allergies. Thorax (2011) 66:1025–6 . doi: 10.1136/thoraxjnl-2011-201019
18 . MetaSUB International Consortium TMI. The metagenomics and metadesign of the subways and urban biomes (MetaSUB) international consortium inaugural meeting report. Microbiome (2016) 4:24 . doi: 10.1186/s40168-016-0168-z
19 . Afshinnekoo E, Meydan C, Chowdhury S, Jaroudi D, Boyer C, Bernstein N , et al. Geospatial resolution of human and bacterial diversity with city-scale metagenomics. Cell Syst. (2015) 1:72–87. doi: 10.1016/j.cels.2015.01.001
20 . Robertson CE, Baumgartner LK, Harris JK, Peterson KL, Stevens MJ , Frank DN, et al. Culture-independent analysis of aerosol microbiology in a metropolitan subway system. Appl Environ Microbiol. (2013) 79:3485–93 . doi: 10.1128/AEM.00331-13
21 . Leung MHY, Wilkins D, Li EKT, Kong FKF, Lee PKH. Indoor-air microbiome in an urban subway network: diversity and dynamics. Appl Environ Microbiol. (2014) 80:6760–70. doi: 10.1128/AEM.02244-14
22 . O’Hara NB, Reed HJ, Afshinnekoo E, Harvin D, Caplan N, Rosen G, et al . Metagenomic characterization of ambulances across the USA. Microbiome (2017) 5:125. doi: 10.1186/s40168-017-0339-6
23 . Gibbons SM, Schwartz T, Fouquier J, Mitchell M, Sangwan N, Gilbert JA, et al. Ecological succession and viability of human-associated microbiota on restroom surfaces. Appl Environ Microbiol. (2015) 81:765–73 . doi: 10.1128/AEM.03117-14
24 . Gilbert JA, Meyer F, Antonopoulos D, Balaji P, Brown CT, Brown CT , et al. Meeting report: the terabase metagenomics workshop and the vision of an earth microbiome project. Stand Genomic Sci. (2010) 3:243–8 . doi: 10.4056/sigs.1433550
25 . Azad MB, Konya T, Maughan H, Guttman DS, Field CJ, Sears MR, et al . Infant gut microbiota and the hygiene hypothesis of allergic disease: impact of household pets and siblings on microbiota composition and diversity . Allergy Asthma Clin Immunol. (2013) 9:15. doi: 10.1186/1710-1492-9-15
26 . Tun HM, Konya T, Takaro TK, Brook JR, Chari R, Field CJ, et al . Exposure to household furry pets influences the gut microbiota of infants at 3–4 months following various birth scenarios. Microbiome (2017) 5:40 . doi: 10.1186/s40168-017-0254-x
27 . O’Connor GT, Lynch S V, Bloomberg GR, Kattan M, Wood RA , Gergen PJ, et al. Early-life home environment and risk of asthma among inner-city children. J Allergy Clin Immunol. (2017) 141:1468–75 . doi: 10.1016/j.jaci.2017.06.040
28 . Birzele LT, Depner M, Ege MJ, Engel M, Kublik S, Bernau C, et al . Environmental and mucosal microbiota and their role in childhood asthma . Allergy (2017) 72:109–19. doi: 10.1111/all.13002
29 . Burgess SL, Gilchrist CA, Lynn TC, Petri WA. Parasitic protozoa and interactions with the host intestinal microbiota. Infect Immun . (2017) 85 : e00101-17. doi: 10.1128/IAI.00101-17
30 . Dominguez-Bello MG, Costello EK, Contreras M, Magris M, Hidalgo G, Fierer N, et al. Delivery mode shapes the acquisition and structure of the initial microbiota across multiple body habitats in newborns . Proc Natl Acad Sci USA. (2010) 107:11971–5. doi: 10.1073/pnas.10026 01107
31 . Neu J, Rushing J. cesarean versus vaginal delivery: long-term infant outcomes and the hygiene hypothesis. Clin Perinatol. (2011) 38:321–31 . doi: 10.1016/j.clp.2011.03.008
32 . National Academies of Sciences, Engineering, and Medicine. Microbiomes of the Built Environment: A Research Agenda for Indoor Microbiology, Human Health, and Buildings. Washington, DC: The National Academies Press (2017). doi: 10.17226/23647 .
33 . McKenzie VJ, Song SJ, Delsuc F, Prest TL, Oliverio AM, Korpita TM, et al . The effects of captivity on the mammalian gut microbiome. Integr Comp Biol . (2017) 57:690–704. doi: 10.1093/icb/icx090
34 . Loudon AH, Woodhams DC, Parfrey LW, Archer H, Knight R, McKenzie V , et al. Microbial community dynamics and effect of environmental microbial reservoirs on red-backed salamanders (Plethodon cinereus). ISME J . (2014) 8:830 –40. doi: 10.1038/ismej.2013.200
35 . Becker MH, Richards-Zawacki CL, Gratwicke B, Belden LK. The effect of captivity on the cutaneous bacterial community of the critically endangered Panamanian golden frog (Atelopus zeteki). Biol Conserv. (2014) 176:199–206 . doi: 10.1016/j.biocon.2014.05.029
36 . Ruiz-Calderon JF, Cavallin H, Song SJ, Novoselac A, Pericchi LR , Hernandez JN, et al. Walls talk: microbial biogeography of homes spanning urbanization. Sci Adv. (2016) 2: e1501061. doi: 10.1126/sciadv.15 01061
37 . Vestergaard DV, Holst GJ, Basinas I, Elholm G, Schlünssen V, Linneberg A, et al. Pig farmers’ homes harbor more diverse airborne bacterial communities than pig stables or suburban homes. Front Microbiol . (2018) 9:870 . doi: 10.3389/fmicb.2018.00870
38 . Lai PS, Allen JG, Hutchinson DS, Ajami NJ, Petrosino JF, Winters T, et al . Impact of environmental microbiota on human microbiota of workers in academic mouse research facilities: an observational study. PLoS ONE (2017) 12 : e0180969. doi: 10.1371/journal.pone.0180969
39 . Ross AA, Doxey AC, Neufeld JD. The skin microbiome of cohabiting couples. mSystems (2017) 2: e00043-17. doi: 10.1128/mSystems.00043-17
40 . Misic AM, Davis MF, Tyldsley AS, Hodkinson BP, Tolomeo P, Hu B, et al. The shared microbiota of humans and companion animals as evaluated from Staphylococcus carriage sites. Microbiome (2015) 3:2 . doi: 10.1186/s40168-014-0052-7
41 . Kraemer JG, Ramette A, Aebi S, Oppliger A, Hilty M. Influence of pig farming on the human nasal microbiota: key role of airborne microbial communities. Appl Environ Microbiol. (2018) 84: e02470-17 . doi: 10.1128/AEM.02470-17
42 . Mosites E, Sammons M, Otiang E, Eng A, Noecker C, Manor O, et al. Microbiome sharing between children, livestock and household surfaces in western Kenya. PLoS ONE (2017) 12: e0171017 . doi: 10.1371/journal.pone.0171017
43 . Barberán A, Dunn RR, Reich BJ, Pacifici K, Laber EB, Menninger HL, et al . The ecology of microscopic life in household dust. Proc Biol Sci . (2015) 282:20151139 . doi: 10.1098/rspb.2015.1139
44 . Akin RD, Heruth DP, Ye SQ, Portnoy JM, Ciaccio CE, Xiong M, et al. House dust bacterial microbiome in smoking and pet owning homes. J Allergy Clin Immunol. (2017) 139: AB86. doi: 10.1016/j.jaci.2016.12.231
45 . Lax S, Smith DP, Hampton-Marcell J, Owens SM, Handley KM, Scott NM, et al. Longitudinal analysis of microbial interaction between humans and the indoor environment. Science (2014) 345:1048–52 . doi: 10.1126/science.1254529
46 . Claus SP, Guillou H, Ellero-Simatos S. The gut microbiota: a major player in the toxicity of environmental pollutants? NPJ Biofilms Microb . (2016) 2:16003 . doi: 10.1038/npjbiofilms.2016.3
47 . Patterson AD, Turnbaugh PJ. Microbial determinants of biochemical individuality and their impact on toxicology and pharmacology. Cell Metab . (2014) 20:761–8. doi: 10.1016/j.cmet.2014.07.002
48 . Spanogiannopoulos P, Bess EN, Carmody RN, Turnbaugh PJ. The microbial pharmacists within us: a metagenomic view of xenobiotic metabolism. Nat Rev Microbiol. (2016) 14:273–87. doi: 10.1038/nrmicro . 2016.17
49 . Abeles SR, Jones MB, Santiago-Rodriguez TM, Ly M, Klitgord N , Yooseph S, et al. Microbial diversity in individuals and their household contacts following typical antibiotic courses. Microbiome (2016) 4:39 . doi: 10.1186/s40168-016-0187-9
50 . Levin AA, Dent JG. Comparison of the metabolism of nitrobenzene by hepatic microsomes and cecal microflora from Fischer-344 rats in vitro and the relative importance of each in vivo. Drug Metab Dispos. (1982) 10:450–4 .
51 . Rickert DE, Butterworth BE, Popp JA, Krahn DF. Dinitrotoluene : acute toxicity, oncogenicity, genotoxicity, and metabolism. CRC Crit Rev Toxicol. (1984) 13:217–34. doi: 10.3109/10408448409003373
52 . Dierickx PJ. Glutathione-dependent cytotoxicity of the chloroacetanilide herbicides alachlor, metolachlor, and propachlor in rat and human hepatoma-derived cultured cells. Cell Biol Toxicol. (1999) 15:325–32 .
53 . Cho Y, Abu-Ali G, Tashiro H, Kasahara DI, Brown TA, Brand JD, et al. The microbiome regulates pulmonary responses to ozone in mice. Am J Respir Cell Mol Biol. (2018) doi: 10.1165/rcmb.2017-0404OC. [Epub ahead of print] .
54 . Jatzlauk G, Bartel S, Heine H, Schloter M, Krauss-Etschmann S. Influences of environmental bacteria and their metabolites on allergies, asthma, and host microbiota. Allergy (2017) 72:1859–67. doi: 10.1111/all.13220
55 . Ridaura VK, Faith JJ, Rey FE, Cheng J, Duncan AE, Kau AL, et al. Gut microbiota from twins discordant for obesity modulate metabolism in mice . Science (2013) 341:1241214. doi: 10.1126/science.1241214
56 . Thomas T, Gilbert J, Meyer F. Metagenomics - a guide from sampling to data analysis. Microb Inform Exp. (2012) 2:3. doi: 10.1186/2042-5783-2-3
57 . Jovel J, Patterson J, Wang W, Hotte N, O’Keefe S, Mitchel T, et al . Characterization of the gut microbiome using 16S or shotgun metagenomics . Front Microbiol. (2016) 7:459. doi: 10.3389/fmicb.2016.00459
58 . Nielsen HB, Almeida M, Juncker AS, Rasmussen S, Li J, Sunagawa S, et al . Identification and assembly of genomes and genetic elements in complex metagenomic samples without using reference genomes. Nat Biotechnol . (2014) 32:822–8. doi: 10.1038/nbt.2939
59 . Knight R, Jansson J, Field D, Fierer N, Desai N, Fuhrman JA, et al. Unlocking the potential of metagenomics through replicated experimental design. Nat Biotechnol. (2012) 30:513–20. doi: 10.1038/nbt.2235
60 . Morgan XC, Huttenhower C. Chapter 12: human microbiome analysis. PLoS Comput Biol. (2012) 8: e1002808. doi: 10.1371/journal.pcbi.1002808
61 . Goodrich JK, Di Rienzi SC, Poole AC, Koren O, Walters WA, Caporaso JG, et al. Conducting a microbiome study. Cell (2014) 158:250–62 . doi: 10.1016/j.cell.2014.06.037
62 . Yang B, Wang Y, Qian P-Y. Sensitivity and correlation of hypervariable regions in 16S rRNA genes in phylogenetic analysis. BMC Bioinformatics (2016) 17:135. doi: 10.1186/s12859-016-0992-y
63 . Wang Q, Garrity GM, Tiedje JM, Cole JR. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Appl Environ Microbiol. (2007) 73:5261–7. doi: 10.1128/AEM.00062-07
64 . Liu Z, DeSantis TZ, Andersen GL, Knight R. Accurate taxonomy assignments from 16S rRNA sequences produced by highly parallel pyrosequencers . Nucleic Acids Res. (2008) 36: e120. doi: 10.1093/nar/gkn491
65 . McDonald D, Price MN, Goodrich J, Nawrocki EP, DeSantis TZ, Probst A , et al. An improved Greengenes taxonomy with explicit ranks for ecological and evolutionary analyses of bacteria and archaea. ISME J. (2012) 6:610–8 . doi: 10.1038/ismej.2011.139
66 . Yilmaz P, Parfrey LW, Yarza P, Gerken J, Pruesse E, Quast C, et al. The SILVA and "All-species Living Tree Project (LTP)” taxonomic frameworks. Nucleic Acids Res. (2014) 42: D643–8. doi: 10.1093/nar/gkt1209
67 . Jordán F, Lauria M, Scotti M, Nguyen TP, Praveen P, Morine M, et al . Diversity of key players in the microbial ecosystems of the human body. Sci Rep. (2015) 5:15920. doi: 10.1038/srep15920
68 . Simpson EH. Measurement of diversity. Nature (1949) 163:688 . doi: 10.1038/163688a0
69 . Shannon CE. A mathematical theory of communication. Bell Syst Tech J . (1948) 27:379–423. doi: 10.1002/j.1538-7305. 1948.tb01338.x
70 . Faith DP. Conservation evaluation and phylogenetic diversity. Biol Conserv . (1992) 61:1–10. doi: 10.1016/0006-3207(92)91201-3
71 . Weiss S, Xu ZZ, Peddada S, Amir A, Bittinger K, Gonzalez A , et al. Normalization and microbial differential abundance strategies depend upon data characteristics. Microbiome (2017) 5:1–18 . doi: 10.1186/s40168-017-0237-y
72 . Lozupone CA, Knight R. Species divergence and the measurement of microbial diversity. FEMS Microbiol Rev. (2008) 32:557–78 . doi: 10.1111/j.1574-6976.2008. 00111.x
73 . Kuczynski J, Liu Z, Lozupone C, McDonald D, Fierer N, Knight R . Microbial community resemblance methods differ in their ability to detect biologically relevant patterns. Nat Methods (2010) 7:813–9. doi: 10.1038 /nmeth.1499
74 . Lozupone CA, Hamady M, Kelley ST, Knight R. Quantitative and qualitative Beta-diversity measures lead to different insights into factors that structure microbial communities. Appl Environ Microbiol. (2007) 73:1576 – 85 . doi: 10.1128/AEM.01996-06
75 . Zaneveld JR, McMinds R, Thurber RV. Stress and stability: applying the Anna Karenina principle to animal microbiomes. Nat Microbiol . (2017) 2:17121 . doi: 10.1038/nmicrobiol.2017.121
76 . Anderson MJ. Distance-based tests for homogeneity of multivariate dispersions. Biometrics (2006) 62:245–53 . doi: 10.1111/j.1541-0420.2005.00440.x
77 . Anderson MJ. Permutational multivariate analysis of variance (PERMANOVA). In: Balakrishnan N, Colton T, Everitt B, Piegorsch W, Ruggeri F, Teugels JL, editors. Wiley StatsRef: Statistics Reference Online . Chichester: John Wiley and Sons Ltd. (2017). p. 1-15 .
78 . Dixon P. VEGAN, a package of R functions for community ecology. J Veg Sci. (2003) 14:927–30. doi: 10.1111/j.1654-1103.2003.tb02228.x
79 . Chakravorty S, Helb D, Burday M, Connell N, Alland D. A detailed analysis of 16S ribosomal RNA gene segments for the diagnosis of pathogenic bacteria. J Microbiol Methods (2007) 69:330–9 . doi: 10.1016/j.mimet.2007.02.005
80 . Hamady M, Knight R. Microbial community profiling for human microbiome projects: tools, techniques, and challenges. Genome Res . (2009) 19:1141 –52. doi: 10.1101/gr.085464.108
81 . Kim M, Morrison M, Yu Z. Evaluation of different partial 16S rRNA gene sequence regions for phylogenetic analysis of microbiomes. J Microbiol Methods (2011) 84:81–7. doi: 10.1016/j.mimet.2010.10.020
82 . Federhen S. The NCBI taxonomy database. Nucleic Acids Res . (2012) 40 :D136–43. doi: 10.1093/nar/gkr1178
83 . Hinchliff CE, Smith SA, Allman JF, Burleigh JG, Chaudhary R, Coghill LM , et al. Synthesis of phylogeny and taxonomy into a comprehensive tree of life. Proc Natl Acad Sci USA. (2015) 112:12764–9. doi: 10.1073/pnas.1423
041112 84 . Balvo ciute M, Huson DH. SILVA, RDP, Greengenes, NCBI and OTT - how do these taxonomies compare? BMC Genomics (2017) 18:114 . doi: 10.1186/s12864-017-3501-4
85 . Langille MGI, Zaneveld J, Caporaso JG, McDonald D, Knights D, Reyes JA, et al. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nat Biotechnol. (2013) 31:814–21 . doi: 10.1038/nbt.2676
86 . Segata N, Waldron L, Ballarini A, Narasimhan V, Jousson O, Huttenhower C. Metagenomic microbial community profiling using unique cladespecific marker genes. Nat Methods (2012) 9:811–14. doi: 10.1038/nmeth . 2066
87 . De Cáceres M, Legendre P, Moretti M. Improving indicator species analysis by combining groups of sites. Oikos (2010) 119:1674–84 . doi: 10.1111/j.1600-0706.2010.18334.x
88 . Knights D, Kuczynski J, Charlson ES, Zaneveld J, Mozer MC, Collman RG, et al. Bayesian community-wide culture-independent microbial source tracking. Nat Methods (2011) 8:761–3. doi: 10.1038/nmeth.1650
89 . Shafiei M, Dunn KA, Boon E, MacDonald SM, Walsh DA, Gu H, et al. BioMiCo: a supervised Bayesian model for inference of microbial community structure. Microbiome (2015) 3:8 . doi: 10.1186/s40168-015-0073-x
90 . Pybus V, Onderdonk AB. Microbial interactions in the vaginal ecosystem , with emphasis on the pathogenesis of bacterial vaginosis. Microbes Infect . (1999) 1:285–92. doi: 10.1016/S1286-4579(99)80024-0
91 . Eden H, Cottee-jones W, Whittaker RJ. The keystone species concept: a critical appraisal introduction: the origins of the concept. Front Biogeogr .(2012)Available online at: http://www.geog.ox.ac.uk/graduate/research /
ecottee-jones.html (Accessed June 19, 2018) .
92 . Herren CM, McMahon KD. Keystone taxa predict compositional change in microbial communities. Environ Microbiol. (2018) 20:2207–2217 doi: 10.1111/1462-2920.14257
93 . Fisher CK, Mehta P. Identifying keystone species in the human gut microbiome from metagenomic timeseries using sparse linear regression . PLoS ONE (2014) 9:e102451. doi: 10.1371/journal.pone.0102451
94 . Bucci V, Xavier JB. Towards predictive models of the human gut microbiome. J Mol Biol. (2014) 426:3907–16. doi: 10.1016/j.jmb.2014.0 3.017
95 . Kearney S, Gibbons S, Erdman S, Alm E. Orthogonal dietary niche enables reversible engraftment of a gut bacterial commensal . bioRxiv[preprint] bioRxiv (2018) 24:1842–1851. doi: 10.1101/275370
96 . Domin H, Zurita-Gutiérrez YH, Scotti M, Buttlar J, Hentschel Humeida U, Fraune S. Predicted bacterial interactions affect in vivo microbial colonization dynamics in Nematostella. Front Microbiol. (2018) 9:728 . doi: 10.3389/fmicb.2018.00728
97 . Olival KJ, Hosseini PR, Zambrana-Torrelio C, Ross N, Bogich TL, Daszak P .Host and viral traits predict zoonotic spillover frommammals. Nature (2017)546:646 –50. doi: 10.1038/nature22975
98 . Hall AB, Tolonen AC, Xavier RJ. Human genetic variation and the gut microbiome in disease. Nat Rev Genet. (2017) 18:690–9 . doi: 10.1038/nrg.2017.63
99 . Brooks AW, Kohl KD, Brucker RM, van Opstal EJ, Bordenstein SR . Phylosymbiosis: relationships and functional effects of microbial communities across host evolutionary history. PLoS Biol . (2016)14 :e2000225. doi: 10.1371/journal.pbio.2000225
100 . Washburne AD, Morton JT, Sanders J, McDonald D, Zhu Q, Oliverio AM , et al. Methods for phylogenetic analysis of microbiome data. Nat Microbiol . (2018) 3:652–61. doi: 10.1038/s41564-018-0156-0
101 . Paradis E, Claude J, Strimmer K. APE: analyses of phylogenetics and evolution in R language. Bioinformatics (2004) 20:289–90 .doi: 10.1093/bioinformatics/btg412
102 . Schliep KP. phangorn: phylogenetic analysis in R. Bioinformatics (2011) 27:592 –3. doi: 10.1093/bioinformatics/btq706
103 . Revell LJ. Phytools: an R package for phylogenetic comparative biology (and other things). Methods Ecol Evol. (2012) 3:217–23 . doi: 10.1111/j.2041-210X.2011. 00169.x
104 . Kembel SW, Cowan PD, Helmus MR, Cornwell WK, Morlon H , Ackerly DD, et al. Picante: R tools for integrating phylogenies and ecology. Bioinformatics (2010) 26:1463–4. doi: 10.1093/bioinformatics / btq166
105 . Orme D. The Caper Package: Comparative Analysis of Phylogenetics and Evolution in R. (2018) Available online at: https://cran.r-project.org/web /packages/caper/vignettes/caper.pdf (Accessed June 20, 2018) .
106 . Harmon LJ, Weir JT, Brock CD, Glor RE, Challenger W. GEIGER : investigating evolutionary radiations. Bioinformatics (2008) 24:129–31 . doi: 10.1093/bioinformatics/btm538
107 . Ho LS, Ané C. A linear-time algorithm for gaussian and non-gaussian trait evolution models. Syst Biol. (2014) 63:397–408. doi: 10.1093/sysbio/syu005
108 . Zaneveld JRR, Parfrey LW, Van Treuren W, Lozupone C, Clemente JC , Knights D, et al. Combined phylogenetic and genomic approaches for the high-throughput study of microbial habitat adaptation. Trends Microbiol .
(2011) 19:472–82. doi: 10.1016/j.tim.2011.07.006
109 . Gianotti RJ, Moss AC. Fecal microbiota transplantation: from clostridium difficile to inflammatory bowel disease. Gastroenterol Hepatol. (2017) 13:209 –13 . Available online at: http://www.ncbi.nlm.nih.gov/pubmed/28546791
(Accessed March 30, 2018 ).
110 . den Besten HMW, Amézquita A, Bover-Cid S, Dagnas S, Ellouze M , Guillou S, et al. Next generation of microbiological risk assessment: potential of omics data for exposure assessment. Int J Food Microbiol . (2017) doi: 10.1016/j.ijfoodmicro.2017.10.006. [Epub ahead of print].